Neue Technologie erleichtert Untersuchung komplizierter Krankheiten
22.12.2022
Mit der 3D-Spatial-Omics-Technologie hat ein Team um Ali Ertürk eine Methode entwickelt, mit der erkrankte Zellen leichter aufgespürt werden können.
22.12.2022
Mit der 3D-Spatial-Omics-Technologie hat ein Team um Ali Ertürk eine Methode entwickelt, mit der erkrankte Zellen leichter aufgespürt werden können.
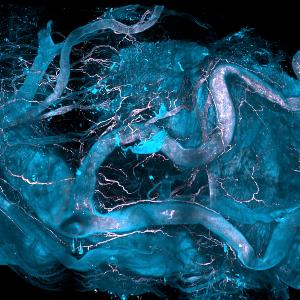
Aortenregionen eines menschlichen Herzens, dessen vaskuläre Plaques mit der DISCO-MS-Technologie analysiert wurden. | © Helmholtz Munich, Harsharan Singh Bhatia und Ali Ertürk
Wie kann eine einzige kranke Zelle in einem intakten Gehirn oder einem menschlichen Herzen aufgespürt werden? Diese Fragestellung gleicht der Suche nach der Nadel im Heuhaufen. Die Teams von Ali Ertürk, Professor für Systembiologie und Technologietransfer an der LMU und Direktor des Institute for Tissue Engineering and Regenerative Medicine (iTERM) bei Helmholtz Munich, und Matthias Mann am MPI für Biochemie in Martinsried bei München haben nun eine neue Technologie namens DISCO-MS entwickelt, die dieses Problem lösen kann. DISCO-MS nutzt Robotertechnologien, um Proteomikdaten von „kranken“ Zellen zu erhalten, die in einem frühen Stadium der Krankheit genau identifiziert werden.
Die meisten Krankheiten verlaufen zunächst asymptomatisch und Betroffene fühlen sich in der Regel noch gut – die Symptome sind noch nicht vorhanden oder noch zu schwach, um sie zu bemerken. Doch im Körper ist bereits eine Veränderung eingetreten: Ein Virus hat begonnen, sich zu vermehren, oder eine abtrünnige Zelle hat sich häufiger geteilt, als sie sollte. Doch wie können diese Veränderungen wahrgenommen werden? Selbst wenn Forscherinnen und Forscher mit Tiermodellen arbeiten, können sie nur selten die spezifische Lokalität des Krankheitsursprungs bestimmen oder die genauen molekularen Veränderungen charakterisieren, die am Fortschreiten der Krankheit beteiligt sind.
Mit der Entwicklung von DISCO-MS durch die Forschungsteams um Ertürk und Mann ist diese Aufgabe viel einfacher geworden. DISCO-MS kombiniert Methoden, die Gewebe von Mäusen und Menschen transparent machen, mit den neuesten Robotik- und Proteomik-Technologien, um ihre molekulare Zusammensetzung zu bestimmen.
DISCO-MS beginnt mit dem sogenannten DISCO-Gewebe-Clearing, das den Mauskörper oder menschliche Organe transparent und damit für die Bildgebung zugänglich macht. Auf diese Weise können fluoreszenzmarkierte Zellen in intaktem Gewebe an bestimmten Stellen mithilfe hochauflösender dreidimensionaler Mikroskopie leicht identifiziert werden.
Nach Identifizierung der interessanten Regionen werden diese mit einer neuen Roboter-Technologie namens DISCO-bot isoliert, die von dem Maschinenbau-Ingenieur Furkan Öztürk, einem Doktoranden in Ertürks Labor, entwickelt wurde. Die robotergestützt entnommenen Gewebe werden für ihre Proteomanalyse mit fortschrittlichen Massenspektrometrie-Methoden verarbeitet, die Andreas-David Brunner entwickelte, ein ehemaliger Doktorand in Manns Labor. Dieser Hightech-Ansatz ermöglicht die vollständige molekulare Charakterisierung jeder gewünschten Geweberegion, die in 3D in ganzen Mauskörpern oder menschlichen Organen identifiziert wurde.
Um die Leistungsfähigkeit der Methode zu demonstrieren, setzten Erstautor Harsharan Singh Bhatia und seine Kollegen DISCO-MS bei einem Mausmodell der Alzheimer-Krankheit (AD) und bei atherosklerotischen Plaques (krankhafte Verhärtungen und Verengungen von Blutgefäßen) im menschlichen Herzen ein.
In den Gewebeproben des Alzheimer-Modells wandte das Team künstliche Intelligenz (KI) an, um die typischen Alzheimer-Plaques in den frühen Stadien der Krankheit zu identifizieren, die mit anderen Methoden bisher nur schwer zu erkennen waren. Anschließende Proteomanalysen der Plaques ermöglichten eine unvoreingenommene und groß angelegte Untersuchung der bei Alzheimer betroffenen Proteine. Dabei wurden neue molekulare Akteure entdeckt, die als Biomarker für die Alzheimer-Krankheit fungieren könnten.
Im menschlichen Herzen interessierten sich die Wissenschaftlerinnen und Wissenschaftler für die Zusammensetzung des Gewebes um atherosklerotische Plaques, die nach der Gewebeentnahme schnell sichtbar wurden. Die KI-Detektion und die robotergestützte Extraktion der Gewebe ermöglichten die Identifizierung dysregulierter molekularer Signalwege in den menschlichen Herzzellen, die mit den Aorten-Plaques in Verbindung stehen. Diese Ergebnisse sind von entscheidender Bedeutung, da sie die Grundlage für potenzielle therapeutische Ziele bilden.
DISCO-MS ist die erste Spatial-Omics-Technologie in intakten 3D-Volumina und beschleunigt somit die Untersuchung komplizierter Krankheiten, von Krebs- bis hin zu Stoffwechsel-Erkrankungen. Da DISCO-MS mit präklinischen und klinischen Geweben arbeitet, ermöglicht die Methode die Untersuchung von Krankheiten in ihren frühesten Stadien und in der Folge auch die Entwicklung potenzieller neuer Therapeutika.
Helmholtz Munich /LMU
Bhatia et al.: Spatial proteomics in three-dimensional intact specimens. Cell 2022